嗨喽~艾薇巴蒂~
还记得一周前的重大事件吗!
北京百迈客生物科技有限公司与牛津纳米孔公司(Oxford Nanopore Technologies,简称ONT)达成长期合作啦!
百迈客本客可以说是肥肠优秀了(围笑)
想必大家对此都饶有兴趣,这里小编先带领大家浏览一下Nanopore技术的成功案例吧!2018年2月7日,Nature Communications在线发表了一篇关于拟南芥基因组组装的文章“High contiguity Arabidopsis thaliana genome assembly with a single nanopore flow cell”。
研究背景
正如大家所知,拟南芥的基因组在在真核生物中有着“金标”之称,但是也仍然存在一定的未知领域。PacBio的出现使高度连续的基因组组合成为可能,但却因读长不够及错误率等方面限制了基因的组装。Oxford Nanopore Technology’s (ONT) MinION 具有超过200 kb的超长读长,5-15%基本错误率。光学图谱与长读长基因组结合才能更好的对基因组序列进行组装。文中使用ONT快速且廉价地发掘了拟南芥结构变异(SV)和数量性状基因座(QTL)。
材料与方法
材料:拟南芥(Arabidopsis thaliana)KBS-Mac-74叶片;
方法:ONT MinION(仅48h!!)+Bionano
研究结果
1、基因组的组装及组装平台的比较
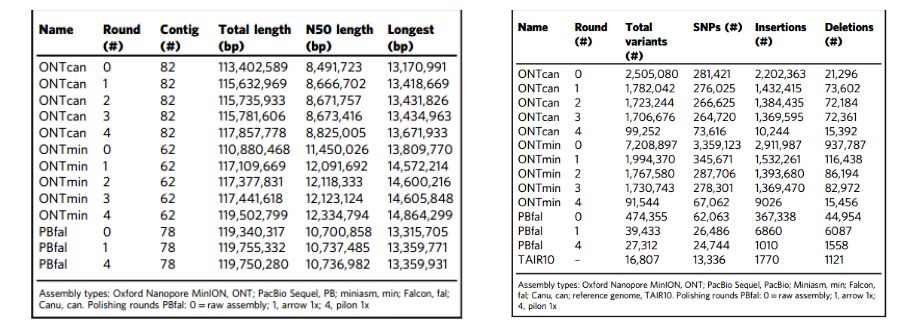
ONT进行一个Cell,共得到3.4 G的数据,平均读长为11.4 Kb(N50=7.5 Kb),最长269 Kb。使用Canu和minimap/miniasm来组装原始的拟南芥ONT fastq数据(分别为ONTcan及ONTmin),并与现在广泛使用的Falcon组装的PacBio数据(PBfal)进行比对(图1)。两种ONT组件都与PBfal相当,并且显示出高度的连续性。ONTmin的组件最小(110.9 Mb),contigs数少(62),但N50(11.5 Mb)长度位于第二,且单个contig最长(13.8 Mb)。
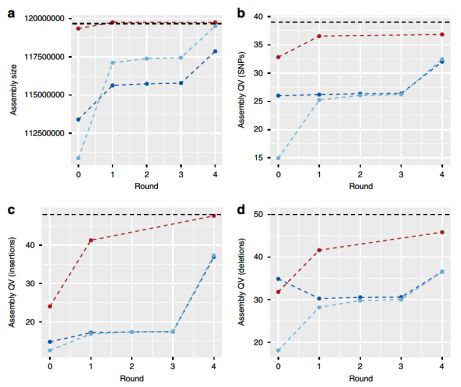
使用三轮racon和一轮pilon对组件进行polish(图1)。发现racon显著地提高了ONT组件的总长。只有第一轮racon和最终pilon的polish对单碱基错配、错误插入和错误删除修正具有强烈影响(图2)。
图1 组装及polish结果比对
图2 单分子组件和TAIR10参考组件的质量比较
2、光学基因组图谱对基础和结构质量的质量控制。
生成265个光学基因组图谱,contigs(cmap)为4.8 Mb,N50=695 Kb。通过光学图谱来评估ONT序列组件的质量。经过racon的polish之后,ONTmin对应到Bionano图谱上的contigs由115.9 Mb增加到118.4 Mb。Pilon的polish改善了两种组装类型,将ONTcan中FP/FN比值从0.33/0.12降到0.01/0.04。
3、超长重复区域的有限组装。
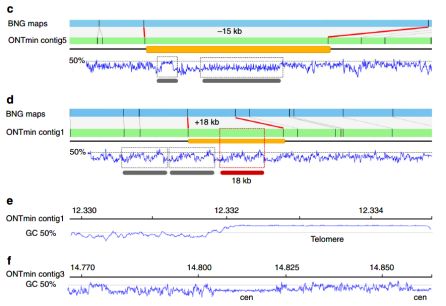
ONTmin 组件包含端粒和着丝粒重复(图3)。PBfal组件具有相似数量的着丝粒和rDNA序列。
筛选完整的和部分基因的基础上使用Quast对TAIR10参考基因组的全基因组比对。结果显示,ONTmin组件具有最高的完整性,紧跟其后的是ONTcan组件。Polish后的ONT组件的基本质量和连续性与同一基因组最先进的PacBio组装相当。
图3 Bionano图谱识别组件分析
4、染色体臂丛揭示性状基因和QTL结构。
将ONTmin组件与拟南芥参考基因组(TAIR10)进行比较,以检测SV。ONTmin组件有4280个SV,总长度为9.5 MB。SV序列的大部分为重复收缩或扩张(58%),其次是插入和缺失(31%)。少数的SV被分类为串联收缩或扩张(11%)。
用CO1-0 At4g30720基因组序列对polish过的KBS-Mac-74 OnTmin组件进行BLAST来分析KBS-Mac-74中的QTL区域。KBS-Mac-74 SG3i区域的更详细的分析显示,它包含四个转座元件片段,与几轮嵌套转座相一致。
以Sanger为基础的BAC测序也不可能精确确定的SG3i区域,在polish后的KBS-Mac-74 ONTmin组件的帮助下,可以在30分钟内得到剖析。
总结
Bionano图再次确认ONT组件的基本质量和连续性,与当前版本的Col-0参考基因组相似甚至超过了它们。具有高基础质量的高度连续的基因组组装具有解决拟南芥种质中功能上重要的存在/缺失多态性的潜力。
超长读取参考基因组提供了获得生物学相关变化的新机会,ONT可以在几天内完成,现在研究人员可以直接处理复杂的基因组区域,不再需要使用以往更费力的方法。基因组组装正成为发掘基因组单一区域上复杂结构变化的最简单的方法!
敲黑板了!
敲黑板了!
醒醒!开始画重点啦!
Oxford Nanopore测序技术:样本处理较为简单,测序周期短,获得数据量高,数据可实时分析;读长更长连续性更高,平均读长高达几十至几百Kb,提高组装指标;能更有效的检测重复序列获得更完整的OTL信息,可进行结构变异检测以及甲基化检测等。为物种表达调控模式的研究提供了新的方法。
Nanopore MinION 测序仪
Nanopore GridION X5测序仪
Nanopore PromethION测序仪
参考文献:
Michael T P, Jupe F, Bemm F, et al. High contiguity Arabidopsis thaliana genome assembly with a single nanopore flow cell[J]. Nature Communications, 2018, 9(1).