原标题:干货|TCR-seq知多少(一)
就在前两个月,小编给大家介绍了HLA这一基因组上的“闹市中心”,可正如北京不仅仅只有三里屯这一块商业繁华区,还有王府井、西单、中关村一般,人类基因组上的“hotspot”还有很多,比如在接下来的两期中将给大家介绍的TCR区域。
参考HLA的介绍,在这一期里我们将先聚焦TCR区域到底是干嘛的、有何特征、有何用这几个基本点来了解这一基因组上的神秘区域。
1、TCR是什么?
T细胞受体(T-cell receptor),简称TCR,是在T细胞表面发现的一种分子[1],T细胞介导的抗原识别依赖于T细胞受体与抗原-主要组织相容性复合体(MHC)的相互作用(图1)。当TCR、抗原肽、MHC结合时,T淋巴细胞通过信号转导被激活,进入后续的免疫应答过程。
TCR是高度多样化的异源二聚体,人类约95%的T细胞表达α链和β链(αβ-TCR,两条链分别由TRA和TRB编码),外周血T细胞(约占1–5%)和粘膜位置T细胞还会表达γδ链(γδ-TCR,分别由TRG和TRD编码)[2],在肿瘤发生和处于某种疾病状态下,αβ-TCR和γδ-TCR比例会发生变化。
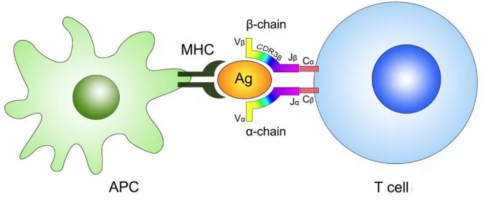
图1 抗原呈递细胞(APC)与T细胞之间的相互作用
2、TCR有什么特征?
TCR复合物的基本功能是引导T细胞识别特定的抗原。面对多种多样的抗原类型如何做到“特异”识别,这就归功于TCR的“高度多态性”了。
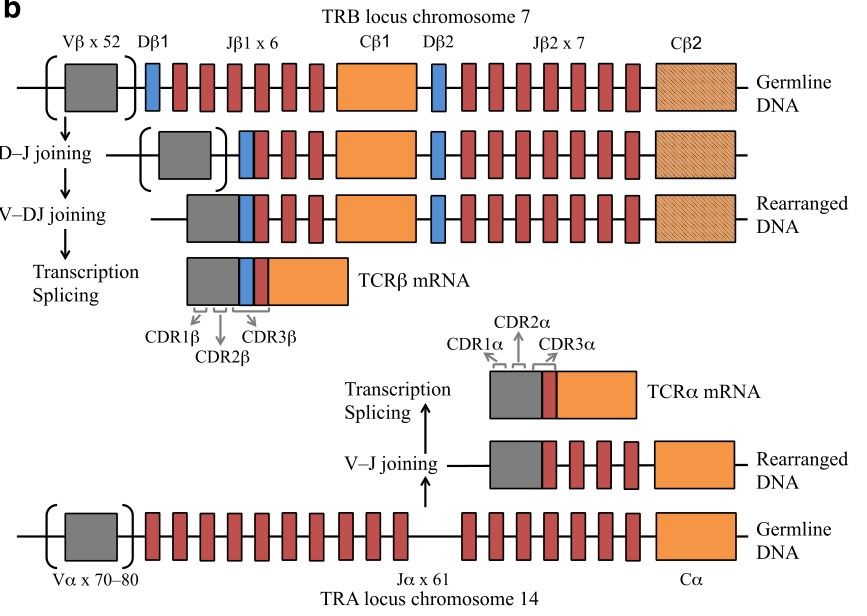
TCR α链和β链由一系列可变(variable,V)和连接(joining,J)基因编码,TCR β和γ链还由高变(diversity,D)基因编码[3,4]。基于RAG1和RAG2重组酶的V-(D)-J重组过程中,V、D、J中一个随机等位基因会与其他区段等位基因重组,形成功能多样化的片段。在V-D、D-J于V-J基因片段之间的连接点处会随机进行核苷酸的添加、缺失。此外,在转录过程中位于重组V(D)J区域的序列以及恒定(Constant, C)区域的基因序列会进行可变剪切,形成存在一定功能差异的转录本(图2)。上述过程会导致超强的重组与结合多样性,产生高可变型、高多样性的T细胞受体,确保对各类抗原进行鉴定。基于数学计算,理论上存在1012-1015种不同的TCR[5],实际人体存在的TCR多样性大约在1013这个数量级,说明看似随机的TCR重组并不是完全随机,可能受到某些机制的约束[6]。
图2 TCR α链和β链V(D)J重组与结合过程
产生TCR多样性的重组过程,是T淋巴细胞在初级淋巴器官——胸腺中的发育早期所特有。每个TCR链都含有3个高度多样性的loop,称为互补决定区域(complementarity determining re-gions, CDR1-3)。CDR1和CDR2由V基因编码,而CDR3由V-J或V-D-J基因结合区域编码,高度多样化。此外,CDR3是TCR直接接触抗原肽的区域,对TCR与抗原肽-MHC复合体的互作起到决定性作用(图1)。正因为如此,CDR3是确定T细胞克隆类型的重要区域,两个T细胞不太可能表达相同的CDR3序列,除非它们来自相同的T细胞克隆[7]。
3、研究TCR有什么用?
既然我们把TCR称为基因组上的另外一个“闹市中心”,可见该区域在人类基因组研究的重要性。那到底TCR可以给我们提供怎样的信息呢?科学家们都在用它进行何种研究和应用呢?下面给大家先进行笼统地介绍,针对TCR在研究中的具体应用,还请关注该主题后面几期的内容哈。
通过TCR profiling(TCR谱)可以分析一个个体T细胞中全部TCR信息,而TCR谱会在疾病的发生与发展中产生巨大变化,这就是为何研究者对特定疾病状态下(如癌症、自身免疫病、炎症以及感染类疾病)患者的免疫组库研究如此感兴趣的原因了。例如,Muraro等[8]使用TCR库分析多发性硬化患者自体干细胞移植对T细胞群的影响;在肿瘤研究中发现细胞毒性T细胞可以在识别肿瘤特异性抗原后杀死肿瘤细胞,一些研究试图通过分析肿瘤浸润淋巴细胞免疫组库确定参与这一过程的特定T细胞克隆类型[9,10]。小编在图3中给各位列举了TCR profiling的热门研究领域。
图3 TCR profiling在人类疾病研究中的应用
参考文献:
[1] Thomas J. Kindt; Richard A. Goldsby; Barbara Anne Osborne; Janis Kuby (2007). Kuby immunology. Macmillan. pp. 223–.ISBN 978-1-4292-0211-4. Retrieved 28 November 2010.
[2]. Presti E Lo, Dieli F, Meraviglia S. Tumor-Infiltrating γδ T Lymphocytes: Pathogenic Role, Clinical Significance, and Differential Programing in the Tumor.
[3] Miles JJ, Douek DC. Price D a. Bias in the αβ T-cell repertoire: implications for disease pathogenesis and vaccination. Immunol Cell Biol. 2011; 89:375–87. doi:10.1038/icb.2010.139.
[4] Burtrum DB, Kim S, Dudley EC, Hayday AC, Petrie HT. TCR gene recombination and alpha beta-gamma delta lineage divergence: productive TCR-beta rearrangement is neither exclusive nor preclusive of gamma delta cell development. J Immunol. 1996;157(10):4293-6.
[5] Davis, M. M. & Chien, Y. H. in Fundamental Immunology (ed. Paul, W. E.) 341–366 (Lippincott-Raven, Philadelphia, 1999.
[6] Laydon DJ, Bangham CRM, Asquith B. Estimating T-cell repertoire diversity: limitations of classical estimators and a new approach. Philos Trans R SocLond Ser B Biol Sci. 2015; 370: 20140291. doi: 10.1098/rstb.2014.0291.
[7] Turner SJ, Doherty PC, McCluskey J, Rossjohn J. Structural determinants of T-cell receptor bias in immunity. Nat Rev Immunol. 2006; 6:883–94. doi:10. 1038/nri1977.
[8] Muraro PA, Robins H, Malhotra S, Howell M, Phippard D, Desmarais C, et al. T cell repertoire following autologous stem cell transplantation for multiple sclerosis. J Clin Invest. 2014; 124:1168–72. doi:10.1172/JCI71691.
[9] Bai X, Zhang Q, Wu S, Zhang X, Wang M, He F, et al. Characteristics of tumor infiltrating lymphocyte and circulating lymphocyte repertoires in pancreatic cancer by the sequencing of T cell receptors. Sci Rep. 2015;5: 13664. doi:10.1038/srep13664.
[10] Gros A, Robbins PF, Yao X, Li YF, Turcotte S, Tran E, et al. PD-1 identifies the patient-specific CD8+ tumor-reactive repertoire infiltrating human tumors. J Clin Invest. 2014; 124:2246–59. doi:10.1172/JCI73639.