转录组广义上指在特定环境(或生理条件)下,一个或一群细胞中所转录出的所有RNA的总和,包括信使RNA(mRNA)、核糖体RNA(rRNA)、转运RNA(tRNA)及非编码RNA;而我们通常所说的转录组则特指mRNA的集合。
随着二代测序价格的不断下降及生信分析技术的不断进步,转录组测序被广泛的应用于生物学研究的方方面面。而“测个序吧”也成了研究者在缺乏明确目标的情况下筛选后续研究方向的一个省时省力,且经济实惠的手段。即使在目前组学研究突飞猛进的情况下,经典转录组也依然占据着极重要的地位(详情请看:你的SCI还缺一个转录组)。然而,对于一些对二代测序技术了解不多的研究者而言,想使用这个方便而强大的工具依然有一定的门槛。

那么,转录组测序到底在做什么呢?到底能做什么呢?实验方案设计上又有哪些讲究呢?下面就跟随小微理清转录组测序的方方面面,为您的研究课题提供一个强有力的工具。
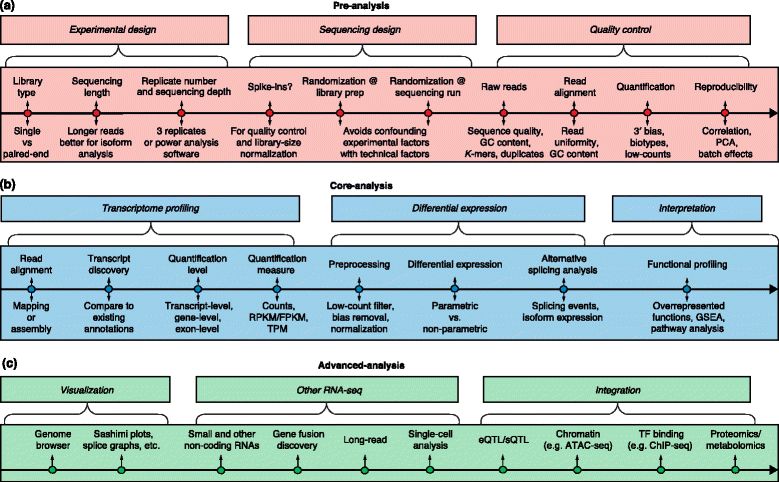
其实早在2016年,佛罗里达大学、加州大学等的研究人员就在Genome Biology上发表了题为“A survey of best practices for RNA-seq data analysis”的review,对经典转录组测序及数据分析作了细致的介绍,目前该综述的累计引用次数已经接近700次,足见转录组测序的火爆程度。我们可以将一个完整的转录组测序的实验流程分为了三部分(图一)。
第一部分是前期分析部分,包括对实验方案的设计、测序方案的设计、以及测序数据的质控。第二部分则是核心分析,包括转录组测序整体评估,基因差异表达分析及功能分析。第三部分是高级分析,这部分需要针对特定的实验目的和需求进行选择,如转录因子的分析、融合基因分析、与其他组学的联合分析等。
图一
前期分析
转录组测序的根本目的在于回答特定的生物学问题,因此一个良好的实验方案的设计是其根本。其中,生物学重复的数量、文库类型及测序深度等因素直接关系到结果的好坏。在这里要尤其强调至少三个以上的生物学重复的重要性。三个以上的生物学重复是进行任何可信的下游数据的统计分析的基础,过少的生物学重复或者没有重复将使分析结果的可信度大大降低。(详情请看:盘点转录组测序问题Top10)。
而由于转录组情况的复杂,选择合适的文库类型也显得极为重要。文库的选择一般考虑两个问题:
01
如何获取mRNA片段?
真核生物成熟的mRNA一般带有polyA尾巴,因此常规的转录组建库流程中通常直接对具有PolyA尾巴的片段进行捕获,这样可以得到纯度较高的mRNA。但是这样的方式对于mRNA的完整度要求较高,发生降解的样本使用这种建库方式会损失一定的转录组信息。另一种获得mRNA的方式则去除在Total RNA中占比最高的rRNA(通常占比超过90%以上),而剩下的RNA中就包括了mRNA(占比为1-2%),这种方式对降解样本的耐受度相对较高,但需要更高的测序数据量,且成本也更高。原核生物由于其mRNA不具有PolyA尾巴,因此只能选择rRNA去除的方式。
02
是否构建链特异性文库?
由于RNA为单链,但普通的转录组文库会同时测到模板链及其反向互补信息,不但无法判断原始的mRNA的方向,同时也会对转录本的定量的准确性产生干扰。而链特异性文库则可以在建库过程中将反向互补序列的文库直接消化掉,不仅保留了原始的mRNA的方向,同时也提高了定量的准确性。
微分基因所有的转录组产品(包括真核有参转录组、真核无参转录组、原核转录组)均已全面升级为链特异性文库。
另一个重要的因素就是测序数据量的大小(即测序深度)。但是最佳的测序数据量并没有一个固定的值,而会因为目标转录组的复杂度的不同而不同。一些人认为5M的mapped reads足够对转录组中的中度及高度表达的转录本进行精确定量了。但是对低丰度的转录本的定量则需要更高的测序深度,而过高的测序深度所带来的转录本的噪声也可能影响定量的准确性。在对转录组的整体评估中,饱和曲线可以较好的评估测序深度是否合适。
通过对以上这些实验设计因素的控制后通过测序就得到了测序数据。我们还需要对原始数据(RAW data)进行质控,包括对低质量的测序数据的去除,接头序列的去除,rRNA序列的去除等。经过质控过滤后得到的数据(clean data)才能进行后续的分析。同时,也可以通过碱基分布、Q20、Q30、rRNA比例等指标初步判断测序质量好坏。
至此,我们得到的clean data就可以进行真正的转录组学分析,来解决我们的实际的生物学问题了。那么需要经过哪些分析流程呢?又可以拿到哪些分析结果呢?请关注转录组测序到底在做什么(二),小微将为您带来有关转录组数据分析方面的详细解答。
参考文献:
1.Conesa A, Madrigal P, Tarazona S, et al. A survey of best practices for RNA-seq data analysis[J]. Genome biology, 2016, 17(1): 13.
2.Hasin Y, Seldin M, Lusis A. Multi-omics approaches to disease[J]. Genome biology, 2017, 18(1): 83.
3.Pimentel H, Bray N L, Puente S, et al. Differential analysis of RNA-seq incorporating quantification uncertainty[J]. Nature methods, 2017, 14(7): 687.
4.Lowe R, Shirley N, Bleackley M, et al. Transcriptomics technologies[J]. PLoS computational biology, 2017, 13(5): e1005457
5.Hardwick S A, Chen W Y, Wong T, et al. Spliced synthetic genes as internal controls in RNA sequencing experiments[J]. Nature methods, 2016, 13(9): 792.
6.Sallam T, Sandhu J, Tontonoz P. Long noncoding RNA discovery in cardiovascular disease: decoding form to function[J]. Circulation research, 2018, 122(1): 155-166.
微分基因为国家大基因中心“基因检测平台”运营方,专注于高通量测序技术,公司凭借国际领先的高通量测序平台,依托独具优势的高通量基因测序和大数据挖掘技术,为各大高校、医院、科研单位以及第三方健康管理服务平台,提供专业的基因检测和数据分析解读服务。2017年,微分基因在国家大基因中心建成2133平方米的洁净分子生物实验室,公司科研团队汇聚了一批国内外优秀的基因组学实验和生物信息分析研究人员。
科技服务部依托公司先进的自动化建库仪、高通量测序仪等实验设备,提供多种测序服务;凭借强大的科研团队,为高校、科研院所等研究单位提供领先的生物信息分析服务。主营业务包括DNA测序、RNA测序、表观组学测序、单细胞测序、ICELL8单细胞表达谱测序、芯片服务、三代全长转录组测序等。