国际生物信息学论坛(International Bioinformatics Workshop,IBW)是由国内外华人生物信息学与计算生物学科学家于2003年共同发起的大规模国际学术交流活动。致力于通过举办高水平学术会议,推动中国生物信息学研究和教育与国际水平接轨。
2019年8月3日,第十四届国际生物信息学论坛(IBW 2019)于北京举办。本次大会由北京大学生物信息中心承办,北京大学数学科学学院/统计科学中心、北京大学分子医学研究所、北京大学医学部基础医学院、北京大学肿瘤医院生物信息中心、蛋白质与植物基因研究国家重点实验室、上海嘉因生物科技有限公司、中软国际科技服务有限公司协办。围绕表观遗传学、基因组学、转录组学、蛋白质组学、系统生物学等前沿科学领域的最新研究进展、技术发展和临床应用,邀请了国内外20位顶尖学者作主题报告。
刘小乐
大会伊始,哈佛大学刘小乐教授为大会致开幕辞。刘小乐回顾了IBW自2003年创办起十六年一路走来的历程。作为中国生物信息学领域的传统盛会,IBW致力于生物信息学的发展和人才培养。大会融汇国内外的专家学者共聚一堂,并设置报告嘉宾与学生座谈环节,促进生物信息领域的交流和创新。
李程
北京大学李程教授代表会议组织方致欢迎辞。李程感谢了IBW论坛委员会各位成员,“龙星课程”的讲师、组织者、助教和学员,以及20位来自国内外的报告人,并向提供服务器支持的华为云致以感谢。作为大会特色之一,9位国际和国内期刊的编辑将在交流会上与参会者进行深入的交流。李程对支持企业、组委会与协办媒体为本届会议做出的付出表示感谢。李程表示,IBW致力于让广大参会者了解课题背后的故事,激发深度交流与灵感碰撞,为生物信息领域的创新研究创造条件。
陈润生
中国科学院生物物理研究所陈润生院士作特约报告。陈润生首先介绍了非编码基因领域的一些最新研究进展。部分非编码基因可以翻译成小肽,这个过程并不是随机的,而是受调控的。研究发现约46%的小肽——像编码基因一样——以AUG作为起始密码子;而对于非AUG起始的小肽,目前正借助离体的翻译体系作进一步研究。非编码RNA存在“过度翻译”现象,比如circRNA可以环绕自身多圈,翻译成更长的肽段。随后,陈润生介绍了基因组上分布于非编码区域中的“超保守序列”,研究表明,这些序列之间的距离也是保守的。这些保守序列先前并未受到广泛关注,它们的功能意义尚待进一步研究,但是这种高度的保守性提示我们这些序列很可能具有潜在的重要意义。在演讲的第三部分,陈润生对DNA计算机的发展做了深入阐述。DNA计算的生化反应是高度并行的,可以实现很快的运算速度,因此如何突破液相反应体系中结果提取的速度限制,成为DNA计算的关键问题。近年来,“DNA折纸”等新技术的发展DNA计算机的应用打下了基础。最后,非编码基因有潜力成为很好的肿瘤标志物或药物靶点,如lncTCF7和lncKdm2b等。
吴建民
王凯
大会第一部分由北京大学肿瘤医院生物信息中心吴建民主持。宾夕法尼亚大学病理系王凯副教授带来了题为《长读长测序在医学基因组学的方法和应用》的报告,基因组上重复序列的变异与一系列疾病相关,如亨廷顿舞蹈症等,但是传统的短读长基因测序技术难以实现准确的鉴定,而长读长测序平台能够更好地鉴定重复串联变异。重复序列鉴定工具RepeatHMM在算法层面做了一系列改进,避免传统方法鉴定重复次数的误差。借助重复序列区域噪音更低的特征,可以利用电信号定位重复序列,王凯向听众介绍了相关的技术模型。接下来,王凯结合具体病例阐述了基于linked-read的长读长测序技术在疾病诊断中的应用,为了克服现有SV鉴定软件的缺陷,王凯团队开发了LinkedSV,可以精准鉴定包括倒位、缺失在内的各种结构变异。
李恒
哈佛大学医学院助理教授李恒的演讲题为《序列比对的艺术》。截止到目前,有100余种序列比对软件被开发出来,李恒对算法的发展历程和未来趋势进行了深入阐述。诞生于高通量测序技术之前的经典的BLAST算法是几乎所有比对软件的始祖。以ELAND、SOAP为代表的第一代短序列比对软件,和目前应用最广泛的第二代软件如Bowtie、BWA、SOAP2等对于较长序列的鉴定速度较慢,第三代短序列比对软件Bowtie 2和BWA-MEM可以达到更长的读长,达到76-300bp。长序列方面,比对软件minimap2是目前应用最广的工具,读长可以达到1kb-250Mbp。李恒认为,新一代短序列比对软件在达到≧150bp的基础上会进一步提升速度,这在大数据时代有着重要意义。与此同时,参考基因组也需要进一步完善,为基因组在科学研究和临床工作中的应用打下更为坚实的基础。
李川昀
臧充之
大会第二部分由北京大学李川昀主持。弗吉尼亚大学助理教授臧充之为听众带来了关于癌症基因组CTCF转录因子结合位点全景图谱的精彩报告。理解蛋白质与基因组之间的相互作用对于探索基因功能和基因与疾病关系至关重要。CTCF作为一种重要的转录因子,它的结合位点在基因组上较为稳定,因此CTCF结合位点的改变可能会带来较为重要的影响。基于多种肿瘤的ChIP-seq数据的研究表明,缺失或新出现CTCF结合位点具有肿瘤特异性;进一步与Hi-C数据和转录组数据结合分析发现,缺失或新出现的结合位点与染色质相互作用以及相应染色质区域的基因表达相关。不断积累的组学大数据可以帮助CTCF结合位点的研究,为我们揭示一系列复杂疾病发生发展的机制。
韩敬东
北京大学定量生物学中心韩敬东研究员的精彩报告为我们对衰老和发育过程的认识提供了全新视角。DNA甲基化钟(DNA Methylation Clock)是目前衰老相关研究的一个热点领域,实现较为准确的年龄预测。与之相似的,基于面部3D影像构建的线性模型也可以实现年龄预测,进一步利用深度学习方法构建的年龄预测模型能够达到更好的预测效果。结合生活习惯数据、血液转录组数据和衰老速率进行分析,可以建立特定生活方式数据(如工作时长、食用冰淇淋、药物使用等)与血液中的标志物及衰老速率的关联。韩敬东向听众分享了利用GEO-seq数据揭示原肠胚形成过程中转录组的动态时空变化的相关研究。
叶凯
西安交通大学叶凯教授以鸦片罂粟基因组和吗啡合成为切入点,探讨了物种进化的不同模式。跳跃式进化和渐进式进化两种现象在物种进化史上同时存在。寒武纪大爆发为跳跃式进化的设想提供了线索。针对这一问题,叶凯团队对罂粟的基因组进行了深入研究,罂粟的分泌物有多种药用作用,包括镇痛、止咳、止泻等。多种活性物质合成通路中的关键酶被陆续鉴定出来,但是与基因组的还尚待阐明。叶凯团队结合短读长高通量测序和长读长测序对罂粟基因组进行了组装,注释了五万余个编码基因。基于组装完成的基因组发现,在罂粟的进化过程中既包含渐进进化也包含跳跃式进化,前者以局部的、小规模的SNP、InDel为代表,后者以全局式的、基因组结构的变异所驱动。
席瑞斌
王艇
大会第三部分由北京大学席瑞斌主持。圣路易斯华盛顿大学医学院的王艇教授为听众分享了三维基因组进化的相关研究。三维结构域主要由序列决定。利用Hi-C技术进行三维基因组研究定位了人和鼠的保守性的拓扑结构区域,王艇指出,约20%转录因子的结合位点来源于转座子,25%的转座子有生化活性,并且具有组织特异性。那么转座子对三维基因组有什么影响?研究发现,具有种属特异性的拓扑域有20%来自转座子,保守的拓扑域有10%是转座子带来的。进一步的基因功能实验表明,转座子在基因调控网络中发挥着一定作用。
马坚
卡耐基梅隆大学计算生物学系的马坚副教授带来了题为《多物种基因组比较的算法》的演讲。基因组具有复杂的三维结构,构成了多种复杂的基因表达调控模式。通过Repli-seq分析不同物种的特定进化模式,鉴定出若干个物种特异的转录因子结合位点。进一步地,基于Ornstein-Uhlenbeck过程的Phylo-HMRF模型,帮助我们在不同物种的Hi-C数据中找到物种间保守的和特异性的结构域,为不同物种间三维基因组的比较提供了新的思路。
徐书华
中科院-马普学会计算生物学伙伴研究所研究组长徐书华教授的演讲以东亚和东南亚人群的融合为主题。相较于欧美人群,东亚人群的大规模基因组学研究比较匮乏。徐书华指出,遗传历程与人类的健康和疾病息息相关。同样的基因突变为什么会带来各异的疾病状态?这背后可能有共享祖源、高发突变、近期基因交流、远古遗传渗入等不同的情况。丝绸之路作为古代人口迁徙的重要通道,其沿线地区人口的融合情况是研究基因流的优良材料。针对汉族人口的基因组分析发现,北方汉族和南方汉族由于和不同地域少数民族的基因交流,表现出一定的差异。徐书华总结说,突变、自然选择、遗传漂变、基因流是形成人种多样性的四大原因。
王欢
徐鹰
大会第四部分由北京大学王欢主持。佐治亚大学徐鹰教授作题为《代谢重编程与肿瘤临床表现》的报告。徐鹰认为肿瘤的转移、耐药等种种外在表现,归根结底来源于肿瘤细胞中的化学反应。一万例以上的不同类型的肿瘤组织和非肿瘤炎症组织基因表达数据的分析,提示肿瘤的代谢重编程现象与某种压力因素相关。通过分析肿瘤细胞的化学反应发现,肿瘤代谢重编程过程都倾向于生成更多的氢离子,而肿瘤细胞pH值仍偏碱性的事实让我们注意到过氧化氢在铁离子催化下生成氢氧根离子的反应的重要性,而过氧化氢和铁离子的存在为免疫细胞和红细胞在肿瘤微环境中的扮演的角色提供了线索。徐鹰强调,将基因组学、转录组学等数据和化学反应结合在一起考虑能够帮助我们提升对于复杂疾病的认识。
李亦学
中国科学院上海生命科学研究院李亦学教授演讲题为《肝癌的精准治疗:从细胞系到病人》。肝癌是全球性的健康挑战,特别是中国,肝癌的发病率和死亡率都相对较高。为了深入研究肝癌的发病机制和分子标记等,细胞系作为一种快速且费用相对低廉的研究模型有着巨大的应用空间。多组学数据分析表明,包含81个细胞系的肝癌细胞系库LIMORE在多个维度上都能够反映原发肝癌的异质性,并且具备癌种特异性。基于LIMORE体系找到肝癌治疗中的重要药物——索拉非尼的潜在伴随诊断生物标志物DKK1,能够较好地预测用药(包括联合用药)的效果。
梁晗
美国MD安德森癌症中心生物信息学与计算生物学系梁晗教授的报告以《站在肿瘤基因组序章的尾声》为题,为听众介绍了在多组学数据的支持下,肿瘤免疫治疗领域的最新研究进展。The Cancer Genome Atlas(TCGA)是一个里程碑式的肿瘤多组学研究项目,通过对涵盖33种癌症超过11000名病人的全方位信息采集和全景式组学描述,使人类对于肿瘤的认识迈上了新的台阶。针对革命性的肿瘤治疗方法——免疫治疗,梁晗提出了三个重要观点:抗体阻断不是检查点抑制的唯一方式,体细胞变异不是突变的唯一来源,基因标志物不是唯一选择。梁晗教授创新性地提出了细胞的平均氨基酸能耗指标ECPAcell对于患者生存可以作为一个独立的预测因子,与TMB等指标结合在一起更可以进一步提升预测能力。
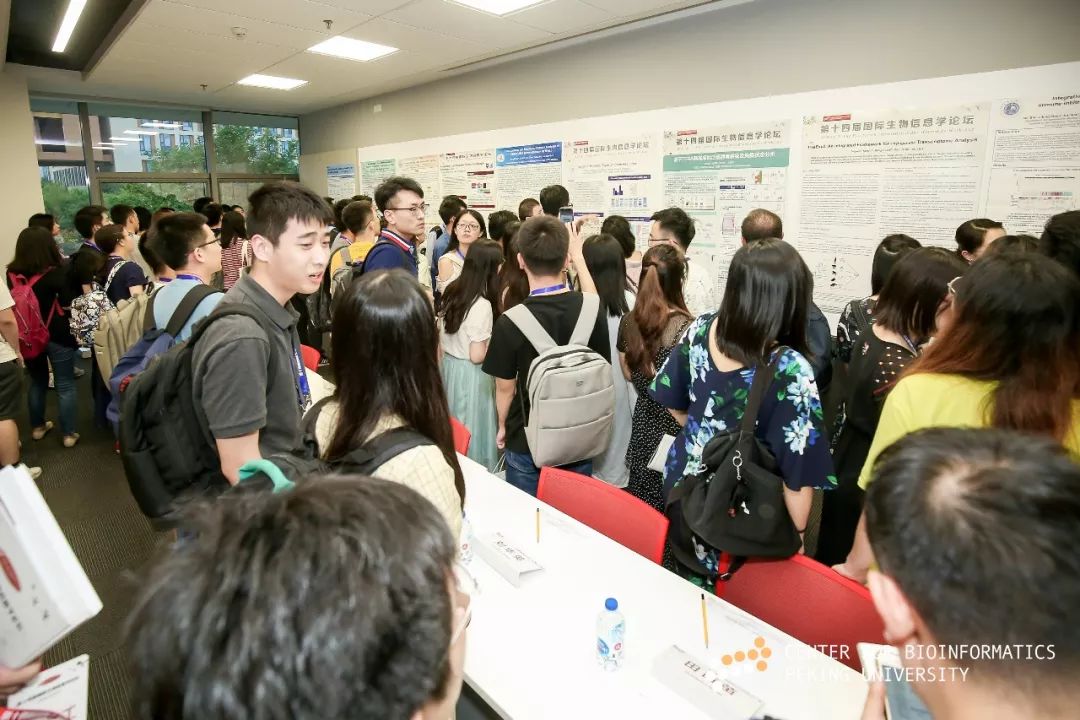
海报展讲
主会场外,大会“海报展讲”环节也吸引了众多参会嘉宾的关注。此外,在“报告人交流会”环节,大会嘉宾与参会的学生面对面地进行了深入交流。针对如何选择研究方向,怎样处理科研中遇到的困难,如何处理与导师的关系等问题,各位报告人结合自己的实际经历,深入浅出、风趣幽默地提出了许多中肯建议,这些经验分享将成为大家学术生涯中的宝贵财富。
报告人交流会
在“期刊编辑交流会”环节,众多参会嘉宾与来自 Genome Biology 、 Genomics, Proteomics and Bioinformatics 、 Journal of Genetics and Genomics 、 Nature Communications 、 National Science Review 、 Quantitative Biology 、 Science Bulletin 期刊的编辑进行了深入的交流。
期刊编辑交流会
大会现场
· END ·