随着基因组测序技术的飞速发展,越来越多的动植物已经完成了全基因组密码的破译,小型基因组(Genome Size<5 Gb)的研究可谓“雨后春笋,势如破竹”,其研究成果发表于众多知名期刊;于此同时,由于三代测序技术(PacBio)成本的大幅度下降,通量>10000倍增加,读长>100倍提升(图1),针对大型基因组(5 Gb≤Genome Size<10 Gb)或超大型基因组(Genome Size≥10 Gb)的组装策略日臻完善,越来越多的科研工作者开展了超大基因组的动植物的研究工作,如裸子植物,两栖爬行动物等。本期小编将为大家介绍下超大基因组的组装策略及研究思路。
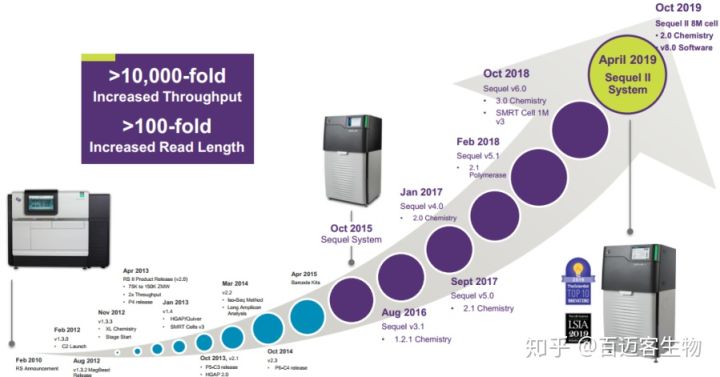
图1 PacBio SMRT测序的发展历史
一、大基因组如何定义?
广义上的大型基因组(Genome Size)通常指基因组大小超过5 Gb,多集中在裸子植物、两栖动物、爬行动物类等。它们往往具有超高的重复序列,还兼具大量的杂合区域。如被子植物(开花植物)基因组大小相差达2400倍(1C = 0.063–148.8 Gb),平均基因组大小为1C=5.7 Gb(图2)。
图2 7542个被子植物中基因组大小(GS)分布的直方图
二、大基因组形成原因?以植物为例,植物DNA C-values数据库(http://www.data.kew.org/cvalues)中提供的植物基因组大小(Genome Sizes,GS)的大规模数据比较分析表明,被子植物(开花植物)的GS多样性显着。植物基因组大小具有多样性主要有两个因素:(1) 多倍体或全基因组复制,引起了植物基因组大小的逐步增加:(2) 拷贝数的变化,可能导致植物基因组大小的变化;DNA重复序列在大多数植物中占据基因组DNA序列的大部分,以几百万个拷贝的形式出现;随着GS的增加,重复DNA序列比例在一定程度增加。
三、大基因组的组装策略PacBio Sequel II平台提供CLR library和HiFi library两种测序模式。其中CLR library可提供超过75 Gb的长片段数据;HiFi library采用CCS方式获取数据,可大幅提升长片段序列的单碱基准确性。Circular Consensus Sequencing (CCS) read: 环形一致性序列,这种一致性序列通过对来自单个ZMW中的subreads进行比对产生。产生的CCS reads不包括或不需要与参考序列比对。产生的CCS reads使用CCS算法需要至少两轮读取来自插入片段的subreads,单条CCS read准确性可达99%。Sequel II System 2.0版本试剂使得HiFi文库的插入片段长度提升至15-20 kb,从而更好的支持基因组从头组装;当使用15 kb HiFi文库时,能够获得更好的变异检测灵敏度和特异性;对于全长转录本以及长片段转录本的检测能力得到提升(图3)。
图3 Pacbio 长片段CCS 建库测序原理图
无论是PacBio CLR测序还是Nanopore测序,均有较高的错误率,常规小基因组物种,在高深度的三代测序数据的基础上(通常为100x),可通过三代原始数据纠错及二代数据polish等方式联合,解决三代数据错误率高,以精准地获得某小基因组物种的基因组密码信息;但针对超大基因组物种,如裸子植物等,一般基因组≥10 Gb,利用常规策略(100x PacBio CLR或Nanopore),由于reads间相互纠错及其耗时,周期长,投入成本巨大且组装质量一般,已经成为了大基因组相关研究中的瓶颈,然而HiFi数据由于高准确性,无需繁琐的纠错过程,30X CCS数据即可完成基因组组装,因此人工组装费用较低、周期较快,组装质量较好,能更好的组装杂合性复杂的物种,使组装的连续性更高,同时结合较长的读长,可以跨越“恼人”的重复区,已成为了越来越多的基因组研究者的最佳选择,如利用HiFi测序技术进行加州红杉基因组组装(图4-5)。
图4 加州红杉基因组组装结果
图5 加州红杉基因组组装结果比较
四、大基因组的研究思路1. 比较基因组研究比较基因组作为基因组研究中的固有思路,在基因组学研究常常加以引用。一般包含系统进化分析,分析时间研究,基因家族收缩与扩张研究,全基因组复制事件WGD研究及共线性分析等。在大型基因组的研究中,同样不例外,可选取10-12个近源种和模式物种,对于其分化地位,物种分化时间,特异收缩扩张基因家族对其生物学功能的影响进行深入研究(图6)。
图6 大蒜基因组组装结果及系统发育分析
2. 基因组重复序列元件分析重复序列是构成真核生物基因组的主要成分。其中转座元件(transposable elements)是DNA重复序列的主要类型,几乎再所有真核基因组中都普遍存在。因此基因组重复序列元件的研究尤其是大型基因组的研究中具有重要的意义与价值,如松柏类的大基因组(20-30 Gb)主要是由于转座子的大爆发导致。(1)重复元件结构分析2020年BioRxiv上报道了重楼属植物七叶一枝花的基因组研究。其预估基因组大小约82.55 Gb,组装获得基因组大小70.18 Gb,该研究中对七叶一枝花的基因组重复序列进行了深入研究,发现在组装获得基因组中,69.53%为重复序列(57.86 Gb),其中62.50%(50.02 Gb)为LTR长末端重复序列。其重复序列含量低于银杏(76.58%),且高于玉米(64.00%)(图7)。
图 7七叶一枝花基因组组装结果及与其它代表性植物基因组特征比较(Genome Size/LTR等)
(2)LTR进化及插入时间分析在宽瓣重楼的研究中,选择银杏、玉米、水稻和拟南芥,使用逆转录酶基因的结构域构建Ty3/ Gypsy和Ty1/ Copia系统进化树。Ty3 /Gypsy可以分为三个进化枝,每个进化枝包含银杏,表明重楼基因组的Ty3/ Gypsy可能是从古老的裸子植物进化而来。发现重楼3个Ty3/ Gypsy进化枝显著扩张,其插入时间显示两次爆发时间(2.2MYA和10.5-11.3MYA),表明所有Ty3/ Gypsy亚型都经历两次扩张Ty1/Copia分成5枝,4枝是重楼特有的,1枝是5个物种共有的(图8)。
图8 两个主要亚型LTR的进化分析及插入时间研究
图8 两个主要亚型LTR的进化分析及插入时间研究
(3)转座子功能研究
大基因组拥有大量转座子,但是这些转座子是否影响该物种的生理生化及生物学功能呢?明确转座子与功能间关系将能对超大基因组存在具有理论意义。如蚯蚓基因组中重复序列LINE2转座元件,显著高比例地插入到蚯蚓早期再生相关的差异基因,同时某些差异表达的LINE2元件和它们的邻近基因拥有极为相似的表达模式,因此LINE2转座元件可能在蚯蚓再生中扮演着重要的调控角色(图9)。
图9 蚯蚓LINE2转座元件功能研究
3. 群体进化研究群体进化研究是指通过获得某物种自然群体各亚群的SNP、InDel等变异信息。然后基于群体变异信息,解析群体的遗传多样性、遗传结构、基因交流情况、物种形成机制以及群体进化动态等生物学问题,从分子层面深入研究该物种的进化历程。同时种群历史动态是分子进化研究的另一核心内容,可用于阐明过去的地质气候变化以及人类活动等历史事件对当前物种分布的影响,同时也有助于制定合理有效的濒危物种保护策略。通过对分子标记在种群历史动态研究中的一些应用案例进行分析,探讨了当前此项研究中存在的问题,揭示了今后的发展趋势。在植物大型基因组的研究中,如裸子植物等,可结合相应的群体材料,对该物种不同群体材料进行深入研究(图10-11)。
图10-11 欧洲银桦群体进化和种群历史动态研究
4. 其它组学研究在大型动植物基因组的研究中,可根据某动植物物种本身的生物学特性,采用相应的生物学技术手段,对该物种生物学特性进行深入研究,如植物(大蒜大蒜素,宽瓣重楼多叶素等,图12-13)、动物(蚯蚓,蝾螈再生机制等,图14)的研究中,可结合代谢组学,单细胞组学,转录组等研究手段,对物种的生物学特性进行深度剖析。
图12 大蒜大蒜素代生物合成通路分析 图13 七叶一枝花多叶素生物合成分析
图14 蚯蚓再生机制单细胞转录组研究
五、参考文献
1.Dodsworth S et al. Genome size diversity in angiosperms and its influence on gene space. ScienceDirect (2015).
2. Sun X et al. A chromosome-level genome assembly of garlic (Allium sativum L.) provides insights into genome evolution and allicin biosynthesis. Molecular Plant (2020).
3. Li J et al. An enormous Paris polyphylla genome sheds light on genome size evolution and polyphyllin biogenesis. BioRxiv (2020).
4. Shao Y et al. Genome and single-cell RNA-sequencing of the earthworm Eisenia andrei identifies cellular mechanisms underlying regeneration. Nature Communications (2020).
5. Salojärvi J et al. Genome sequencing and population genomic analyses provide insights into the adaptive landscape of silver birch. Nature Genetics( 2017).











