导读
在空间背景下分析细胞转录组对于人们理解肿瘤进展和治疗耐药性机制至关重要。近年来,空间转录组学(ST)发展迅速,基因覆盖范围从少数靶点到全基因组,细胞分辨率从亚细胞到多细胞。作为ST方法的一个关键分支,基于分子条形码的原位捕获策略能够在完整组织中无偏倚地捕获整个转录组,但其可能检测来自不同谱系多个细胞的混合信号,无法有效区分恶性细胞和真正未知的细胞类型。
另一种基于反卷积细胞组分的策略是生成与同一肿瘤样本ST数据配对的单细胞转录组测序数据(scRNA-seq),但单细胞实验通常需要新鲜样本。此外,即使是新鲜样本,scRNA-seq也可能捕获不到某些类型的细胞(如中性粒细胞)。
近日,美国国家癌症研究中心姜鹏团队在Nature Communications发表了题为“Estimation of cell lineages in tumors from spatial transcriptomics data”的文章,并详细介绍了团队自主开发的计算模型——SpaCET(Spatial Cellular Estimator for Tumors)。SpaCET可以预测肿瘤ST数据中每一个空间位置的细胞成分,解决了肿瘤异质性、组织密度变化、免疫细胞完整性和细胞谱系间共线性的挑战。经验证,SpaCET在横跨7种癌症类型的8个肿瘤ST数据集中的预测性能优于其他方法。此外,SpaCET还可提供肿瘤进展相关潜在细胞间互作分析功能。

文章发表在Nature Communications
肿瘤空间转录组中的细胞谱系分解
SpaCET模型通过三个步骤来预测肿瘤空间转录组数据中的细胞谱系和细胞间相互作用。首先,SpaCET基于拷贝数改变(CNA)和常见肿瘤类型的恶性转录组基因特征图谱来估计恶性细胞分数。其次,SpaCET在统一的线性回归模型下对非恶性细胞组分进行反卷积,并使用来自不同癌症类型的scRNA-seq数据集构建参考表达谱,计算非恶性细胞(如免疫细胞、基质细胞等)的比例。最后,整合细胞共定位分析和配体-受体共表达分析,SpaCET能够揭示肿瘤微环境中潜在的细胞间相互作用。
图1. 肿瘤空间转录组学中细胞组分和相互作用的推断。来源:Nature Communications
通过模拟ST数据进行性能验证
为评估SpaCET的性能,研究团队使用双盲组织病理学注释对模拟和真实ST数据进行反卷积,计算了模拟中分解的细胞分数与实际细胞混合比之间的相关性和均方根误差,并将其作为评估指标。
研究团队收集了已发表的10个scRNA-seq数据集,使用来自不同细胞谱系的数千个单细胞构建了一个全面的细胞图谱,并对数据集内部和数据集之间进行验证,以评估SpaCET对合成ST数据的分解性能。结果显示,在大多数细胞类型分解的数据集内,SpaCET都实现了较高的预测精度。此外,在特定细胞谱系中,如恶性细胞、癌症相关成纤维细胞、中性粒细胞细胞和浆细胞,SpaCET同样具有较高的预测精度。
研究团队还将SpaCET与几种具有代表性的ST和细胞类型反卷积方法进行比较,评估其对合成ST数据的分解性能。在模拟环境中,SpaCET在主细胞类型和亚细胞类型层面均优于其他方法。此外,研究团队还计算了所有方法的运行时间和内存消耗,结果显示SpaCET属于高效算法。
图2. 基于模拟ST数据的SpaCET性能评估。来源:Nature Communications
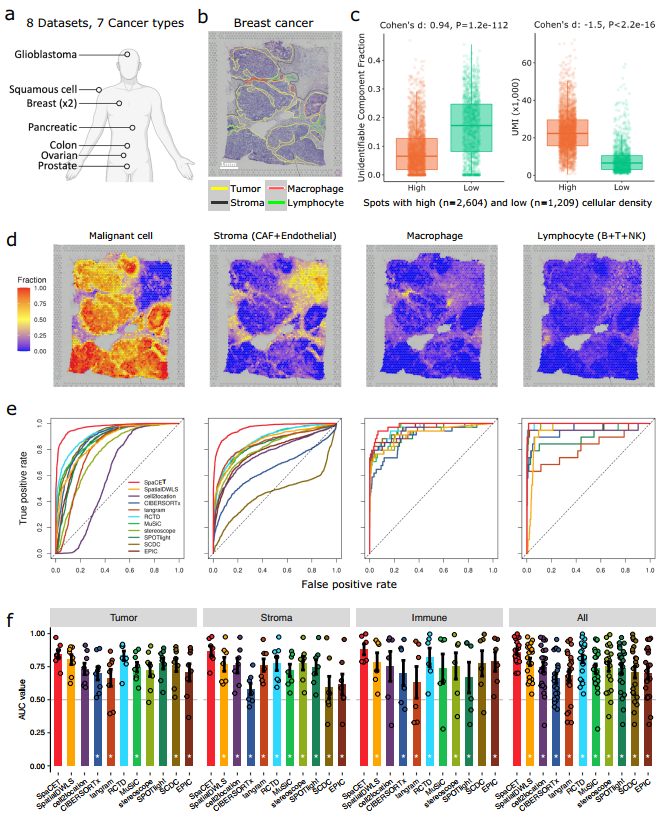
通过真实ST数据和双盲组织病理学注释进行验证
研究团队利用SpaCET对8个真实肿瘤ST数据集进行了分解,涵盖7种肿瘤类型,数据横跨了3个不同分辨率的测序平台,包括Slide-Seq、10X Visium、以及初代的ST技术。
研究团队根据ROC曲线比较了SpaCET与以往方法的预测精度。SpaCET无需恶性细胞特征,并具有内置的正常细胞参考,但大多数已有方法需要单细胞参考进行反卷积,因此研究团队收集了现有的scRNA-seq数据并构建了泛癌单细胞参考图谱。对比结果显示,SpaCET总体上能够更准确地预测癌细胞、间质细胞以及免疫细胞的空间分布。SpaCET在恶性细胞、基质细胞和巨噬细胞中的空间预测性能优于其他方法;在淋巴细胞中,SpaCET与其他方法(如Stereoscope和RCTD)性能相当。
图3. 基于真实ST数据和双盲组织病理学注释验证SpaCET性能。来源:Nature Communications
SpaCET揭示空间环境下细胞间的相互作用
肿瘤细胞的相互作用在癌症进展和治疗耐药性中起着关键作用。scRNA-seq数据可以通过分析跨细胞类型的配体-受体共表达来揭示细胞间通信,但会丢失邻近信息,可能会得出假阳性结果。SpaCET的反卷积结果能够在空间背景下进行细胞相互作用分析。
为从ST数据中识别细胞间相互作用,SpaCET采用了两步法:评估细胞共定位;然后进行配体-受体(L-R)共表达分析。基于估计的细胞分数,研究团队计算了ST点上细胞类型对之间的Spearman相关性,细胞类型对的强正相关表明其细胞共定位。
接下来,研究团队通过分析ST点内L-R的相互作用以证明细胞相互作用。每个ST点的L-R评分表明了每个位点配体-受体的相互作用强度,而非细胞类型之间的特定相互作用。因此,SpaCET进一步对每个细胞类型对进行了L-R评分的富集分析。例如,对于乳腺肿瘤组织中CAF和M2细胞的共定位,SpaCET将所有ST点分为四类:CAF - M2共定位、CAF或M2占主导地位以及其他;并发现CAF-M2共定位的点比CAF/M2占主导地位的点具有更高的L-R得分。
图4. SpaCET识别乳腺肿瘤中的细胞间相互作用。来源:Nature Communications
结 语
综上所述,研究团队提出了SpaCET,以解决肿瘤ST数据中分解细胞的难题。研究表明,SpaCET在ST数据上优于现有的反卷积方法。SpaCET基于肿瘤类型的拷贝数改变和表达变化的基因模式来估计癌细胞分数,在空间分布预测精度和运行效率方面都优于其它方法。随着临床研究中空间转录组数据的不断积累,SpaCET将为多种致癌过程提供更深层面的机制见解,并为当前抗肿瘤治疗的瓶颈提供新的治疗方案。
参考文献:
1. Ru, B., et al. Estimation of cell lineages in tumors from spatial transcriptomics data. Nat Commun 14, 568 (2023).
2. Grisanti Canozo, et al. Cell-type modeling in spatial transcriptomics data elucidates spatially variable colocalization and communication between cell-types in mouse brain. Cell Syst. 13, 58–70.e5 (2022).
3. Barkley, D. et al. Cancer cell states recur across tumor types and form specific interactions with the tumor microenvironment. Nat. Genet. 54, 1192–1201 (2022).