21年前,第一版人类基因组图谱正式发布,引爆了当年的生命科学研究领域。
2001年2月,人类基因组计划(Human Genome Project, HGP)的研究成果“人类基因组的最初测序和分析”由Nature率先发表。随后Science发表了Celera基因组公司的“人类基因组测序”研究成果。这两项相互竞争又各自独立完成的人类基因组草图的发表,被认为是人类基因组计划的里程碑。
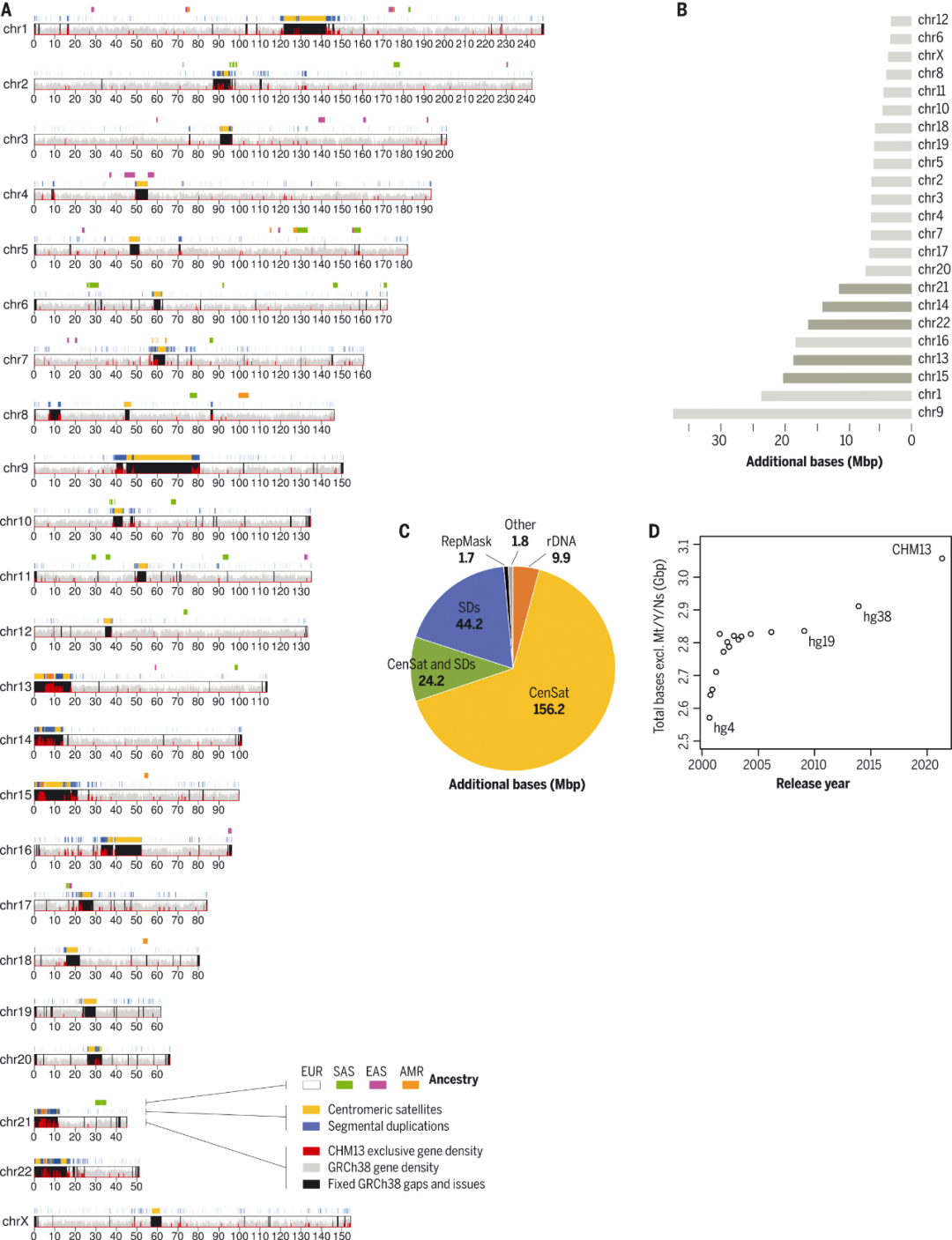
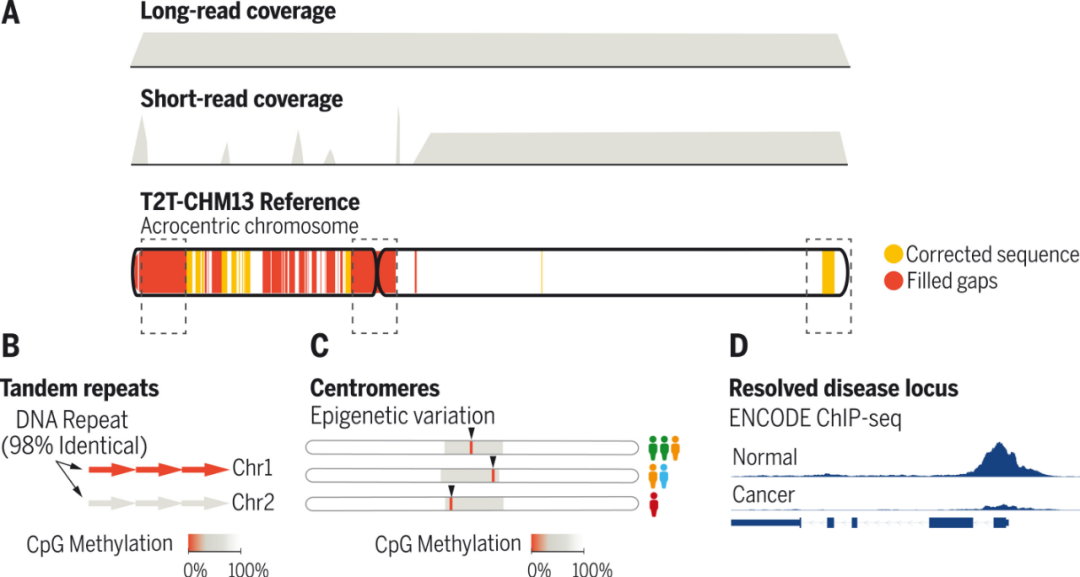
遗憾的是,由于当时的技术限制,部分人类基因组区域未能实现测序解读,人类基因组图谱并不完整,并且存在一些错误。经过后续多次更新,现在的人类基因组版本为GRCh38.p14(GRCh38)。但GRCh38缺失了人类基因组约8%的序列,有169段重要的重复序列未能成功拼接;相当一部分序列难以分析组装。此外,具有重要生物功能的染色体近端着丝粒短臂、着丝粒和多个重复的常染色质区域也未解析。

当地时间3月31日~4月1日,Science以特刊形式发表了端粒到端粒(T2T)联盟的研究成果,报告了最新的人类参考基因组(T2T-CHM13),包括人类所有22条常染色体和X染色体的无缝组装。该成果完成了人类基因组计划中8%尚未解决的具有挑战性的任务,主要通过对葡萄胎(单倍体)的长读长测序实现,为人类基因组增加了∼200Mb的遗传信息,相当于一个完整的染色体。该特刊共发布6篇文章,详细介绍了人类近全基因组从端粒到端粒组装分析完成、表观遗传转录基因组分析、重要重复序列的解读分析等,纠正了以往基因组序列的许多错误,并解锁了人类基因组中结构最为复杂的一些区域。
图:每个长条代表一条染色体的线性可视化,左边显示的是染色体编号。红色片段表示T2T联盟解析的此前缺失的序列。来源:T2T联盟
通过解析以前无法测序和比对的序列区域,这些区域大多由高度重复的序列组成,绘制的参考基因组可以对着丝粒卫星重复、转座元件和片段重复进行详细的描述。基因组图谱的绘制,包括以前发表的相关研究,解决了人类遗传多样性的各个方面,包括人类与灵长类动物的进化比较。此外,基因组图谱可用于识别着丝粒内和着丝粒之间甲基化丰度的变化如何不同,以及表观遗传学如何影响重复序列的转录。
此次发布的系列研究成果梳理了T2T参考基因组如何影响生物医学相关变异的检测,以及驱动决定人类特征的基因组区域的进化。虽然还有很多基因信息有待挖掘,但T2T参考基因组提供了另一个有助于继续探索的基准,可以深入研究完整自我背后的遗传学。
The complete sequence of a human genome
自首次发布以来,人类参考基因组只覆盖了部分基因组,一些重要区域尚未完成解析。T2T联盟解决了剩余8%的基因组分析,提供了一个完整的包含30.55亿碱基对的人类基因组图谱——T2T-CHM13,包括除Y染色体外所有染色体的无缝组装,纠正了此前参考图谱中的错误,并引入了近2亿个碱基对序列,包含1956个基因预测,其中99个预测是蛋白质编码基因。完整的区域包括所有的着丝粒卫星阵列,近端重复区域,以及5个端中心染色体的短臂,解锁这些复杂基因组区域可以进行变异和功能研究。
图:完整的T2T-CHM13人类基因组组装概述。
A complete reference genome improves analysis of human genetic variation
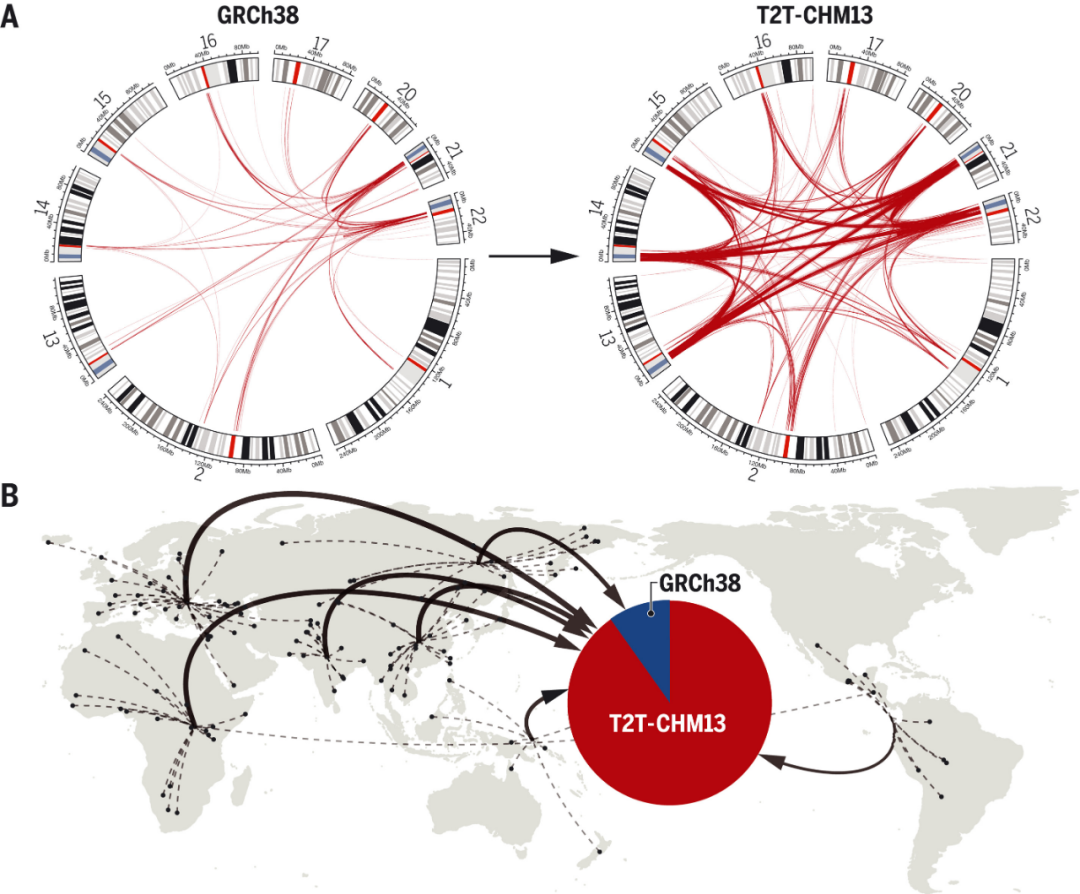
与此前的CHM13基因组图谱相比,T2T-CHM13图谱增加了近2亿个碱基对,纠正了数千个结构错误,并解锁了人类基因组中最复杂的区域,用于临床和功能研究。该研究展示了该参考图谱如何普遍地改进全球3202个短读长测序和17个长读长测序不同样本的read mapping和变异调用。研究团队在此前未解决的序列区域确定了每个样本中的数十万个变异,展示了T2T-CHM13图谱在进化和生物医学发现方面的应用前景。同时,该研究消除了每个样本中数以万计的假阳性变异,包括使269个医学相关基因检测的假阳性降低了90%以上。由于这些变异检测的改进,联合群体和功能基因组资源,T2T-CHM13图谱被定位为取代GRCh38作为人类遗传学的参考图谱。
图:T2T-CHM13的基因组特征和资源。
Segmental duplications and their variation in a complete human genome
高度相同的片段重复(SDs)在疾病和进化中有着非常重要的作用,也是人类参考基因组(GRCh38)最后被完全测序的区域之一。利用完整的人类T2T-CHM13图谱,研究团队提出了人类SD组织的全面视角。SDs约占新增序列的三分之一,在全基因组范围的估值从5.4%增加到7.0%(2.18Mbp)。268个人类基因组的分析表明,91%的未解决的T2T-CHM13 SD序列(68.3Mbp)更好地代表了人类拷贝数变异特征。通过比较人类(n=12)和非人类灵长类动物(n=5)基因组的长片段组装,研究团队系统重建了生物医学相关和重复基因的进化和结构单倍型多样性。上述分析揭示了结构杂合模式和人类与其他灵长类动物之间SD组织的进化差异。
图:更完整的片段重复信息提高了基因分型。
Complete genomic and epigenetic maps of human centromeres
现有的人类参考基因组序列几乎完全排除了着丝粒内部和周围的重复序列,这些序列约占基因组的6.2%(189.9兆字节),这限制了人们对其组织、进化和功能(包括促进染色体的正常分离)的理解。现在,为全面地描述着丝粒周围及内部重复序列,T2T联盟发布了一个完整的、从端粒到端粒的人类参考基因组序列——T2T-CHM13。重复序列区域的详细图谱揭示了数兆碱基结构的重排,包括活跃的着丝粒重复序列。着丝粒相关序列的分析揭示,着丝粒的位置与其周围DNA的分层重复扩增进化之间存在较强关联。此外,对不同个体X染色体着丝粒的比较表明,在这些复杂且快速进化的区域内存在着结构、表观遗传和序列的高度差异。
图:无间隙组装阐明着丝粒的进化
From telomere to telomere: The transcriptional and epigenetic state of human repeat elements
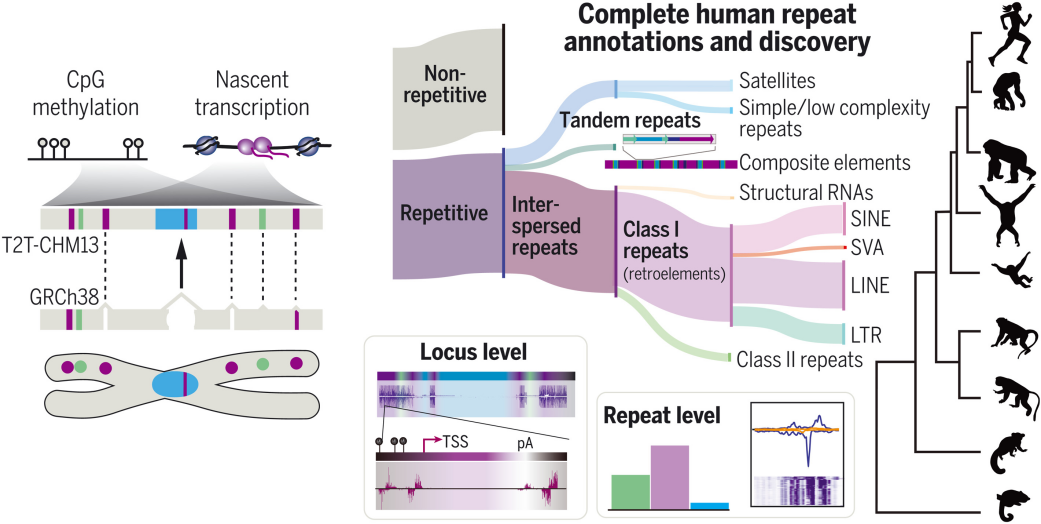
移动元件和重复基因组区域是独特的谱系特异性基因组,是指纹个体基因组形成的主要原因。对这类重复元件(包括在基因组更复杂区域中发现的重复元件)的综合分析,需要完整的线性基因组组装。为此,研究团队从头重复测序和注释了T2T-CHM13。研究团队确定了以前未知的卫星阵列,扩展了重复和移动元件的变体及家族目录,表征了复杂复合重复序列的类别,并定位了逆转录元件转导事件。此外,研究团队还检测了新生转录并绘制了CpG甲基化谱,以定义人类转录活性逆转录元件的结构,包括着丝粒中的逆转录元件结构。这些数据扩展了人们对塑造人类基因组重复区域的多样性、分布和进化的认知。
图:人类参考基因组T2T-CHM13支持重复注释和发现。
Epigenetic patterns in a complete human genome
人类参考基因组新版本T2T-CHM13解析了基因组中的复杂区域,包括重复和同源区域。研究团队发现了在以往的高分辨率表观遗传学研究中从未被解析的序列。该序列是一个包含整个近端着丝粒染色体短臂、基因家族扩增及重复类的多样化集合,精确映射了CpG甲基化(32800个CpG)、DNA可及性和短片段数据集(166058个未解析的染色质免疫沉淀测序峰),提供了此前未识别或校正基因之间存在活动的证据,并揭示临床相关的同源特异性调控。通过对来自6个不同个体着丝粒CpG甲基化的进一步探索,该研究还对着丝粒定位的变异性进行了评估。综上所述,该研究为研究人类基因组中最复杂的区域提供了框架,为表观遗传调控提供了新见解。
图:完整的人类基因组表观遗传特征。
同时,Science还发布了遗传学家Deanna Church教授的评述文章“A next-generation human genome sequence”。Deanna M. Church在评述中表示,21年前,Celera基因组公司和人类基因组计划(HGP)发布了两个人类基因组序列的初始版本。这些图谱是不完整的,且存在一些错误。但绘制人类基因组参考图谱的价值是显而易见的,因此HGP图谱在过去的十年中不断更新完善。遗憾的是,人类基因组图谱仍存在许多缺点。此次发布的新图谱将对人类基因组分析产生重大影响,并意味着向组装代表所有人类的基因组模型迈出的重要一步,这将更好地支持个性化医疗、人群基因组分析和基因组编辑。